また投稿タイトルがイマイチしっくりこなかったのだけど、やりたかったことといえば割とランダムなXY座標点の強度(x,y,I)で描かれている画像を、例えばx,y座標0.1刻みずつといった定点のグリッドの強度座標に直したかったということ。
データは以下のような感じ。ある程度格子に近い座標にデータはあるけれど、それぞれの座標は定点に乗っているわけではないという感じ。
X Y I
-0.87 -0.84 3.5
-0.32 -0.83 10
-0.09 -0.85 4.5
0.08 -0.87 5.3
0.35 -0.88 6.2
0.90 -0.84 3.1
-0.85 -0.74 4.5
-0.31 -0.73 15
-0.13 -0.75 2.2
0.07 -0.77 3.6
0.35 -0.78 11
0.88 -0.74 5.2
... ... ...
-0.86 0.84 35
-0.33 0.83 12
-0.12 0.85 53
0.11 0.87 12
0.32 0.88 18
0.94 0.84 19
大概の場合このままのランダム座標で処理して困ることもないのだけど、別の座標系に変換したいってときにグリッドデータから変換したほうが都合が良かったってことがあって、座標点をグリッドの定点に書き換えるってことを試したのがこの投稿。
とりあえずtkinterでdataの読み込み。
tk = Tk()
tk.withdraw()
print ('select a data files')
filenames = tkfd.askopenfilenames(filetypes= [('text','*.dat')], initialdir='./DATA')
tkms.showinfo('file paths are',filenames)
tk.destroy()
data = [0]*len(filenames)
basename = [0]*len(filenames)
for i in range(len(filenames)):
data[i] = np.loadtxt(filenames[I])
basename[i] = os.path.basename(filenames[i]+str(i)+'.txt')
scipyのinterpolate.griddataで強度座標の変更
使ったのはscipyのinterpolate.griddataを試してみることに。
展開するためのグリッド座標点を準備する。numpyのmgridなりmeshgridなりを使って。
#meshgridで。グリッドの粗さはdataに合わせて
xi = np.linspace(-0.1,0.1,100)
yi = np.linspace(-0.1,0.1,100)
x_grid,y_grid = np.meshgrid(xi,yi)
#mgridならこんな感じ
x_grid,y_grid = np.mgrid[-0.1:0.1:101j,-0.1:0.1:101j]
読み込んだdataを並び換えてから、griddataを使う。interpolateのオプションは3種類cubic, linear, nearest。
for i in range(len(filenames)):
coords=np.vstack((data[i][:,0].flatten(),data[i][:,1].flatten())).T
grid_data1 = griddata(coords,data[i][:,2].flatten(),(x_grid,y_grid),method='cubic')
grid_data2 = griddata(coords,data[i][:,2].flatten(),(x_grid,y_grid),method='linear')
grid_data3 = griddata(coords,data[i][:,2].flatten(),(x_grid,y_grid),method='nearest)
できた座標をpyplotのscatterを使ってプロット。
r_min = 0
r_max = -1
v_min=0
v_max=50
fig,ax=plt.subplots(2,2)
ax[0,0].scatter(data[i][r_min:r_max,0],data[i][r_min:r_max,1],c=data[i][r_min:r_max,2],cmap=cm.gist_rainbow,marker='s',vmin=v_min,vmax=v_max,s=plotsize)
ax[0,0].title.set_text('original')
ax[0,1].scatter(x_grid,y_grid,c=grid_data1,cmap=cm.gist_rainbow,marker='s',vmin=v_min,vmax=v_max,s=plotsize)
ax[0,1].title.set_text('cubic')
ax[1,0].scatter(x_grid,y_grid,c=grid_data2,cmap=cm.gist_rainbow,marker='s',vmin=v_min,vmax=v_max,s=plotsize)
ax[1,0].title.set_text('linear')
ax[1,1].scatter(x_grid,y_grid,c=grid_data3,cmap=cm.gist_rainbow,marker='s',vmin=v_min,vmax=v_max,s=plotsize)
ax[1,1].title.set_text('nearest')
#なんか良い方法が見つからなかったんだけど軸設定はもう少しスッキリかける?
ax[0,0].set_xlim(-0.1,0.1)
ax[0,1].set_xlim(-0.1,0.1)
ax[1,0].set_xlim(-0.1,0.1)
ax[1,1].set_xlim(-0.1,0.1)
ax[0,0].set_ylim(-0.1,0.1)
ax[0,1].set_ylim(-0.1,0.1)
ax[1,0].set_ylim(-0.1,0.1)
ax[1,1].set_ylim(-0.1,0.1)
plt.savefig('FIGURE/TEST/'+'griddata'+basename[i]+'orig.png')
それで画像はこんな感じ。
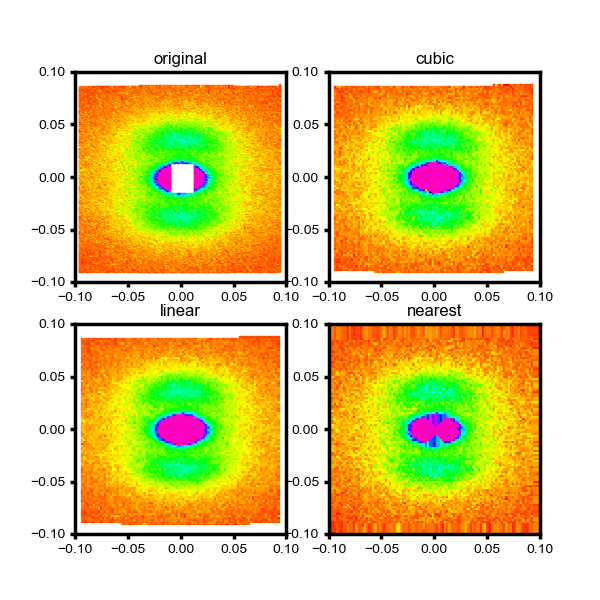
見た感じcubicとlinearはそれなりに良さそうに見える。実際にこんな座標変換が許されるかはデータと目的次第でしょうので、必要なデータ処理にバイアスがかかってないかはチェックする必要があるでしょう。
さて、これでだいたい良いって場合もあるかもしれないけれど、このdataの場合だと中心のマスク部分の座標点が取り除かれている処理がされているので、その座標点分がintnerpolateで高強度に染められてしまうという問題?が。
numpyのmeshgridをマスクする
そもそもscipyの違うモジュールでやるって手もあるんだろうけど、今回は単純にグリッドの座標にマスクを追加して修正。
#mask xy grid for center
x1,x2,y1,y2 = 0.0125, -0.0101, 0.0138, -0.0152
rect = (x_grid < x1) & (x_grid>x2) & (y_grid < y1) & (y_grid > y2)
x_grid=x_grid[~rect]
y_grid=y_grid[~rect]
もしくはnanに変更するとか。
x_grid[rect]=np.nan
y_grid[rect]=np.nan
これで画像はこんな感じに。ちょっとマスクのサイズ間違ってるっぽいけど、まあ良いでしょう。
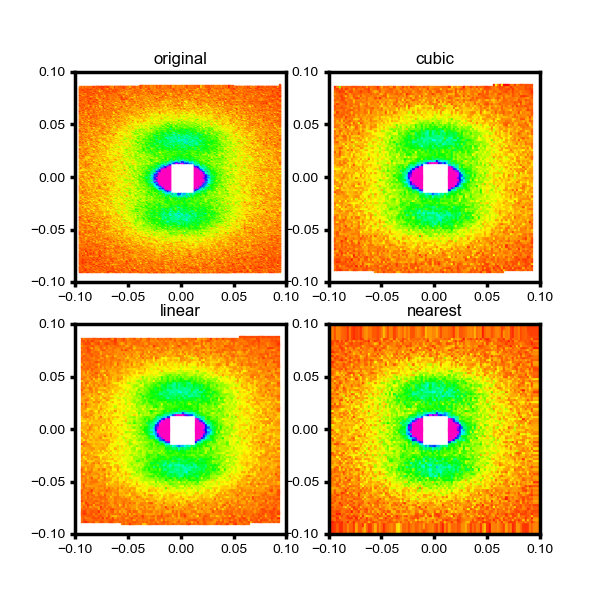
という感じで一応定点グリッドに座標を移し替えられました、という投稿でした。
関連記事
1. pythonのまとめ
D